Ученые из Объединенного института биоэнергетики (JBEI) помогают изменить эту ситуацию с помощью новой технологии ENTRAP-seq, которая позволяет одновременно анализировать тысячи регуляторов транскрипции у растений. Регуляторы транскрипции — это белки, которые влияют на экспрессию гена подобно регулятору яркости света; они могут полностью его отключить или изменить количество определенного продукта, производимого клеткой, модулируя частоту транскрипции ДНК в РНК.
Большинство улучшенных признаков, которые мы наблюдаем у современных сельскохозяйственных культур и видов растений для биотоплива, являются результатом модуляции транскрипции; например, тысячи лет назад древние земледельцы выводили природные варианты пшеницы с более высокой экспрессией генов, контролирующих размер зерна. Ученые в области сельскохозяйственных технологий хотели бы лучше использовать эти генетические переключатели, однако наше понимание их ограничено, несмотря на обширные исследования геномов растений.
«Даже для хорошо изученных растений, о которых мы много знаем, какие гены контролируют какие процессы и во многих случаях, как именно работает ген, неясно, как изменить экспрессию генов для внесения полезных изменений», — сказал Саймон Аламос, научный сотрудник Калифорнийского университета в Беркли и JBEI, научно-исследовательского центра биоэнергетики Министерства энергетики США, управляемого Национальной лабораторией имени Лоуренса Беркли (Berkeley Lab).
Аламос является соавтором исследования, описывающего ENTRAP-seq, опубликованного в журнале Nature Biotechnology. «Мы хотим иметь возможность использовать собственные регуляторы транскрипции растений, а также белки с такой активностью из других организмов, например, растительные вирусы, но до сих пор у нас не было способа предсказать, что они делают, или быстро их протестировать», — говори Аламос.

Разработка и оптимизация анализа для параллельного измерения активности транс-действующих элементов в растительных клетках. Источник: Nature Biotechnology (2025). DOI: 10.1038/s41587-025-02880-w
ENTRAP-seq заполняет эту неудовлетворенную потребность, уменьшая размеры экспериментов, которые ранее требовали целого растения или целого листа, до одной клетки. В этом процессе используется бактерия, поражающая растения, для вставки в лист последовательностей ДНК многих потенциальных белков-регуляторов транскрипции, а также кода для сконструированного целевого гена.
Ученые создают библиотеку из тысяч бактерий, каждая из которых содержит инструкции для одного варианта белка. Затем бактериальная библиотека вводится в лист, и каждая бактерия переносит свой генетический материал в отдельную растительную клетку, так что тысячи различных вариантов производятся разными отдельными растительными клетками.
После того как растительная клетка синтезирует эти компоненты, варианты белка способны включать или выключать молекулярные переключатели внутри клеточного ядра, сигнализируя о наличии у них активирующих или подавляющих свойств. Если белок обладает активирующими свойствами, экспрессируется специально разработанный ген, способный связываться с магнитными метками. Затем ученые могут использовать магниты для физического выделения клеток, которые произвели целевой белок, секвенировать ДНК внутри них и сопоставить результаты со списком потенциальных белков, которые они ввели.
После разработки подхода команда продемонстрировала его беспрецедентную скорость в реальных исследованиях. Они использовали ENTRAP-seq для изучения регуляции генов в транскрипционном активаторе, который, как известно, опосредует экспрессию гена, контролирующего цветение у арабидопсиса, растения, используемого в качестве модельного организма в ботанических исследованиях. Они создали 350 мутантных версий белка и быстро проверили, какие из них могут регулировать время цветения в большую или меньшую сторону.
«Этот эксперимент занял всего несколько недель. В отличие от него, в предыдущем исследовании наша команда охарактеризовала активность 400 регуляторов транскрипции растений, на что двум специалистам потребовалось два года работы в режиме полной занятости, используя традиционный, грубый подход тестирования каждого элемента по отдельности», — сказал ведущий автор Патрик Ши, заместитель вице-президента отдела сырья и директор по проектированию растительных биосистем в JBEI.
Раскрывая панель управления
Ши и его команда разработали потенциальные варианты активаторов для этого исследования, используя существующую модель искусственного интеллекта, которая идентифицирует последовательности ДНК, способные кодировать белки, активирующие гены, из любого организма.
«Предсказания модели позволили нам сосредоточить экспериментальные усилия на наиболее информативных белках в тысячах геномов», — сказал соавтор Лукас Вальдбургер, аспирант-исследователь из Калифорнийского университета в Беркли и Института биотехнологий и инженерии (JBEI).
Однако точность этой модели ограничена нехваткой обучающих данных, поскольку, как ученые знают не понаслышке, методы исследования регуляторов транскрипции раньше были чрезвычайно медленными. С помощью ENTRAP-seq исследователи смогут быстро создавать огромные наборы данных, которые позволят усовершенствовать эти модели.
Более совершенные модели позволяют исследователям быстро сканировать геном растения и обнаруживать, где закодированы все генные переключатели — информация, которая практически отсутствует для многих видов. Затем, замыкая петлю положительной обратной связи, ENTRAP-seq поможет ученым изучать влияние естественных и искусственно созданных вариаций этих белков для создания версий, улучшающих желаемые признаки.
«Мы ищем все переключатели и хотим знать, являются ли они переключателями «включено» или «выключено»? И насколько высоко и насколько низко можно их опустить? Каталогизация всего этого станет огромным шагом вперед. Затем, возможно, мы найдем важный переключатель для выращивания более крупных растений, повышения урожайности или повышения устойчивости культур к стрессам, и, наконец: можем ли мы выкрутить этот регулятор яркости на максимум?» — сказал Ши, который также является доцентом Калифорнийского университета в Беркли и научным сотрудником в области бионаук лаборатории Беркли.
Источник: Национальная лаборатория имени Лоуренса в Беркли. Автор: Алия Ковнер.
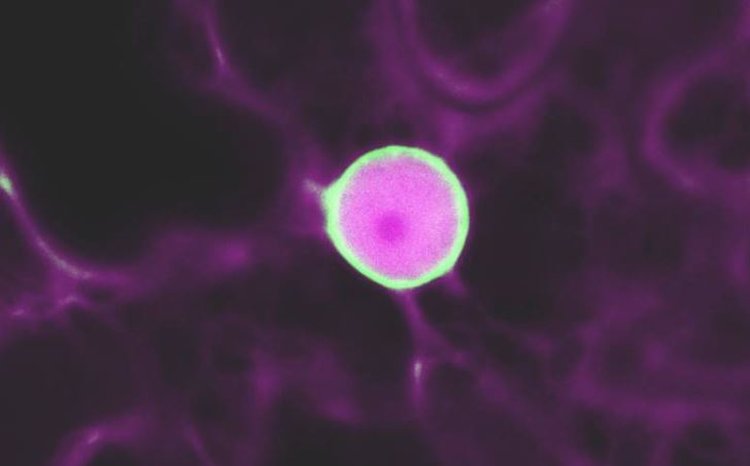
На фото - изображение одной из клеток листа, полученное с помощью конфокальной микроскопии. Пурпурный цвет обозначает репортер транскрипции, представляющий собой красный флуоресцентный белок, показывающий, что активатор транскрипции, введенный в эту клетку, активировал синтетический целевой ген. Зеленый цвет обозначает зеленую флуоресцентную метку на целевом гене и прикрепленную к нему магнитную метку, накопившуюся в ядре клетки. Автор изображения: Саймон Аламос/Лаборатория Беркли.